酿酒微生物岗位
刘延琳团队
摘 要:目的:建立一种快速从酿酒酵母突变库中筛选出低产乙醇酵母菌株的方法。方法:以转录因子spt15随机突变酿酒酵母文库为研究模型,以高生物量产率作为低产乙醇酿酒酵母的筛选标记,利用24孔板对spt15突变酵母文库进行初筛,对初筛获得的目标菌株,再以乙醇产量作为筛选标记进行复筛,最终获得乙醇合成能力较低且生物量较高的酿酒酵母菌株。
结果:(1) 利用易错PCR技术构建了spt15随机突变文库,并将其转化至酿酒酵母YS59(Saccharomyces cerevisiae YS59),获得了S. cerevisiae YS59 spt15突变文库;(2)经过初筛,获得了与对照菌株相比,生物量产率≥70%的14株S. cerevisiae YS59spt15突变菌株;(3)通过复筛,筛选出了编号712的低产乙醇突变菌株,其乙醇合成量降低了28.1%;(4)通过序列比对,发现spt15-712上有9个碱基发生了突变,分别是A161G、T426C、T462C、T493A、A506G、T546C、T622C、T658C、A750T9。结论:成功建立了一种高通量筛选低产乙醇酿酒酵母的方法,并利用这种方法筛选出了乙醇合成量降低28.1%的低产乙醇突变菌株。
关键词:酿酒酵母;生物量产率;低产乙醇;spt15;突变
随着全球气候的变暖,葡萄的糖含量在逐年升高,导致葡萄酒乙醇含量也随之增加,在过去的20年中,酒度平均升高了2%(v/v)。较高的酒度给葡萄酒带来辛辣等各种不良感官影响。因此,在葡萄酒酿造过程中适当降低乙醇的合成,成为葡萄酒工业亟待解决的问题。利用物理化学方法诱变,或者利用基因工程策略改造葡萄酒发酵用酿酒酵母,而后对酿酒酵母突变库进行筛选,获得乙醇合成能力弱化的酿酒酵母菌株,将是一种降低葡萄酒酒度最为经济和简便的方法。为了从酿酒酵母突变库中筛选出低产乙醇的酵母菌株,需要对突变库中所有菌株进行发酵实验并检测其乙醇产量。
目前,常用的乙醇检测技术,包括:(1)仪器法,如气相色谱法仪和高效液相色谱仪法,这两种方法检测限高、速度快,但是使用费用普遍较高;(2)物理/化学法,如莫尔氏盐法、快速氧化法、密度瓶法,这类方法操作复杂,效率低;(3)酶法,利用醇脱氢酶(ADH)催化,使乙醇与氧化型烟酰胺腺嘌呤二核苷酸(NAD)反应,然后测定还原型烟酰胺腺嘌呤二核苷酸(NADH)荧光强度变化率,从而得出其酶促反应速率,对应乙醇的浓度制成标准曲线,样品中乙醇含量由标准曲线计算求得,该法乙醇含量检出限较高,为4.0×10-6mol/L,但酶法同样存在操作复杂效率低等问题。
由此可见,由于缺少乙醇高通量检测方法,在从上述酵母突变库中筛选低产乙醇酵母菌株时,将面临筛选工作量巨大且效率低等问题。为此,需要开发一种新型的,能够快速从酿酒酵母突变库中,筛选出低产乙醇酵母菌株的方法。
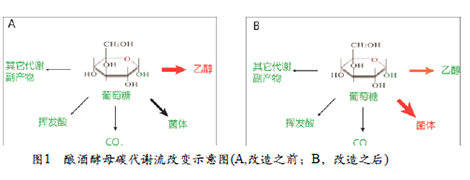
在同等发酵体系下,根据物质守恒的原则,拥有较高生物量合成能力的酿酒酵母菌株,意味着有更多的碳源(糖)用于菌体的合成(图1),而更少的碳源(糖)用于乙醇等其他代谢产物的合成。本研究以转录因子spt15随机突变酿酒酵母文库为研究对象,利用24孔板,以高生物量产率作为低产乙醇酿酒酵母的筛选标记,对spt15突变酵母文库进行初筛,对初筛获得的潜在目标菌株,再进一步通过模拟葡萄汁发酵并检测其乙醇合成能力,最终将获得乙醇合成能力较低且生物量较高的酿酒酵母菌株。本研究所采用的筛选策略将大幅度提高筛选效率和降低筛选成本。
1 材料与方法
1.1 材料与设备
1.1.1 菌种和质粒
Saccharomyces cerevisiae YS59,本研究室保藏;Escherichia coli JM109感受态细胞,大连宝生物公司;质粒pY16TEF1,江南大学刘立明教授惠赠。
1.1.2 主要试剂
总DNA提取试剂盒、质粒提取试剂盒、DNA胶回收试剂盒、酵母快速转化试剂盒,美国Omega生物技术公司;Diversify® PCR RandomMutagenesis Kit,Clontech;蛋白胨、酵母膏、酵母浸粉、酸水解酪蛋白,北京奥博星生物技术有限责任公司;YNB,北京索莱宝科技有限公司;-Ura do supplement,Clontech;葡萄糖、硫酸铵、泛酸(分析纯),四川西陇化工有限公司。
1.1.3 主要设备
MJX智能霉菌培养箱,宁波江南仪器厂;ELX800酶标仪,美国BioTek公司;MB100微孔板恒温振荡器,杭州奥盛仪器有限公司;SBA生物传感分析仪,山东省科学院生物研究所。
1.1.4 培养基
(1)LB培养基;(2) YPD培养基;(3) SD-Ura培养基;(4)模拟葡萄汁
1.2 试验方法
spt15随机突变文库→spt15随机突变酿酒酵母文库→以生物量产率为指标初筛→高生物量产率菌株→以乙醇产率为指标复筛→低产乙醇菌株→spt15突变位点检测
1.2.1 spt15基因的PCR扩增
根据NCBI数据库中S. cerevisiae S 2 8 8 c s p t 1 5 基因序列( G e n eAcces s ion No. :NC_001137)
和质粒pY16 TEF1序列, 利用primer premier 5.0软件设计引物spt15 P1/P2。引物委托上海生工合成。上游引物spt15 P1:5’-ATAGGGACCTAGACTTCAGG- 3’。下游引物spt15 P2:5’-GACCTCCCATTGATATTTAAG -3’。以50μL PCR 反应体系进行扩增,反应体系为:

1.2.2 spt15随机突变质粒文库的构建
按照Diversify® PCR RandomMutagenesis Kit(Clontech CodeNo.630703)使用说明,对spt15基因进行随机突变。其中,引物为:Primer F:5’- TAGAACTAGTGGATCCATGGCCGATGAGGAACGTTTAAAG’- 3’;Primer R:5’- GCTTGATATCGAATTCTCACATTTTTCTAAATTCACTTAGCACAG- 3’

1.2.3 酵母质粒的转化
按照Q u i c k & E a s y Ye a s tTransformation Mix (Cat. No.631851)试剂盒说明操作,将spt15随机突变质粒文库, 转化到S .cerevisiae YS59中,得到spt15随机突变酵母文库。
1.2.4 spt15随机突变酿酒酵母文库菌株的发酵试验
(1) 突变文库菌株的活化
将spt15随机突变酿酒酵母文库菌株分别接种于无菌24孔细胞培养板。24孔板的每孔加入1.5mLSDUra培养基,接种50μL菌液,置于微孔板摇床,400rpm,25℃活化24h。
(2) 高生物量产率酵母菌株的筛选
将活化好的突变菌株,按每孔50μL接种量,接种于每孔装有1.5mLSD-Ura培养基的无菌24孔细胞培养板。每个菌株均有四个重复,25℃条件下进行静置发酵。待发酵结束后检测每个菌株的OD560nm值。
(3) 低产乙醇目标菌株发酵能力的验证
根据1.2.4(2)的实验结果,挑选出OD560nm值较高的菌株,利用SD-Ura培养基活化后,按照2% 的接种量将其接种至装有250mL模拟葡萄汁的500mL三角瓶中,置于25℃条件下静置发酵,并在发酵过程中检测糖的消耗。待发酵结束后,检测OD560nm值、乙醇含量和挥发酸含量。
1.2.5 spt15基因突变碱基的检测
spt15基因的PCR扩增条件同
1.2.1,将PCR产物送上海生工公司
测序。将测序结果同S. cerevisiaeS 2 8 8 c s p t 1 5 基因序列( G e n eAccession No.:NC_001137)进行比对,找到突变碱基。
1.3 测定方法
酿酒酵母生物量(OD值)的测定,利用ELX800酶标仪对样品在560nm下进行OD值检测。葡萄糖的测定,按照SBA-40E型生物传感分析仪使用说明进行测定;总还原糖的测定,采用DNS法;挥发酸的测定,参照GB15038-2006T进行;乙醇的测定,SBA生物传感分析仪测定。
2 结果与分析
2.1 spt15随机突变酿酒酵母文库的构建
提取酿酒酵母Y S 5 9 的总DNA,利用PCR 扩增出spt15基因。将PCR扩增产物与pY16TEF1分别进行Eco RI和Bam HI双酶切,并连接。对连接产物进行测序,结果表明spt15基因连接正确,将其命名pY16TEF1-spt15。利用易错PCR试剂盒对pY16TEF1-spt15重组质粒中的spt15基因进行随机突变,得到spt15基因突变文库△spt15。将△spt15与pY16TEF1进行连接,获得质粒突变库pY16TEF1-△spt15。将重组质粒pY16TEF1-△spt15转化E. coli JM109,随机挑选20个转化子,提取质粒并进行spt15基因测序,结果显示spt15基因突变位点个数在2~10个不等,且突变位点和碱基无偏好性,从而说明spt15随机突变文库构建成功。
利用Q u i c k & E a s y Ye a s tTransformation Mix (Cat. No.631851)试剂盒,将spt15随机突变质粒文库,转化到S. cerevisiae YS59中。将转化液均匀涂布在SD-Ura固体培养基上,30℃培养96h左右,获得转化子(图2)。将所有转化子用液体SD-Ura培养基培养后保藏,从而获得S. cerevisiae YS59 spt15突变文库。

2.2 高生物量产率酿酒酵母菌株的筛选
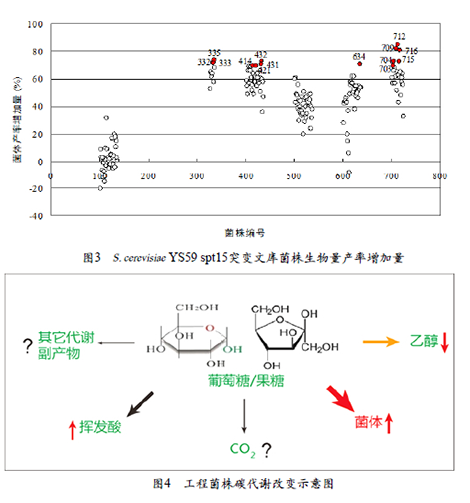
利用S D - U r a 液体基进行S .cerevisiae YS59 spt15突变文库菌株的发酵,结果显示,与对照菌株S.cerevisiae YS59-pY16(含pY16TEF1)相比较,大部分菌株的生物量产率均有所增加,其中,编号为421、414、703、634、431、332、333、715、432、704、335、716、709和712,共14个菌株的生物量产率菌均大于70%,分别达到70%、70%、70%、71%、71%、72%、72%、73%、73%、73%、74%、81%、82%和85%(图3)。
2.3 高生物量酿酒酵母菌株的模拟葡萄汁发酵特征
为了验证上述所筛选出的14株菌株的产乙醇能力是否降低,将其分别进行葡萄模拟汁发酵试验(表1)。由表1可见:(1)乙醇产量均不同程度降低。乙醇产量低于9度的分别是634、703、712号菌株,分别比对照S. cerevisiae YS59-pY16降低了21.5%、27%和28.1%,其中的712号菌株的乙醇产率最低,达到0.327 g/g糖;(2)虽然各菌株均能顺利完成发酵,但是所用时间差异较大。704在26天完成发酵,而716需要49天才能完成发酵;(3)挥发酸均显著升高。所有测试菌株的挥发酸产量均超过3.0 g/L,远高于GB15037-2006中规定的不超过1.2g/L的要求。这说明,随着乙醇合成能力的减弱,来自于底物葡萄糖/果糖的碳,在减少流向乙醇合成途径的同时,增加了向挥发酸合成途径的流动,比如可能增强了乙酸合成途径(图4)。
2.4 低产乙醇菌株中spt15基因突变碱基的鉴定
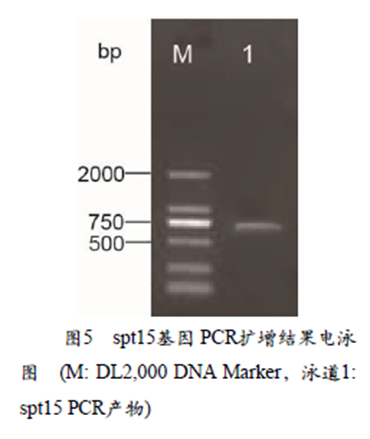
提取712#酿酒酵母质粒(pY16-spt15-712),以spt15 P1/P2为上下游引物,按照1.2.1的方法,对spt15基因进行PCR扩增(图5),将扩增产物(spt15-712)进行切胶回收后,送上海生工公司进行测序。利用Vector NTI软件(10.3.0版)将测序结果与未突变的spt15基因序列(GeneAccession No.:NC_001137,spt15-ck)进行比对( 图6),发现spt15-712上有9个碱基发生了突变,分别是A161G、T426C、T462C、T 4 9 3 A 、A 5 0 6 G 、T 5 4 6 C 、T622C、T658C、A750T。

3 结论
本文建立了一种高通量筛选低产乙醇酿酒酵母的方法。目前,虽然可以使用气相色谱法、密度瓶法、酶法等进行乙醇含量的检测,但是,这些方法在进行乙醇高通量检测时,均会存在效率不高等问题。为此,本文以S. cerevisiae YS59spt15突变文库菌株为研究对象,以高生物量产率作为低产乙醇酿酒酵母的筛选标记进行筛选。通过检测S. cerevisiae YS59 spt15突变文库菌株发酵结束后的OD560nm值,筛选出编号为332、333、335、414、421、431、432、634、703、704、 709、712、和715的高生物量产率菌株。将上述菌株分别利用葡萄模拟汁进行发酵,并检测其乙醇产量,结果显示,与对照菌株S .cerevisiae YS59-pY16相比较,以高生物量产率所筛选出来的菌株,其乙醇产量降低了7.1%~28.1%,其中,712号菌株乙醇产量降低最多,达到28.1%。对712号菌株的质粒pY16-spt15-712测序,发现spt15基因上共有9个碱基发生了突变,分别是A161G、T426C、T462C、T 4 9 3 A 、A 5 0 6 G 、T 5 4 6 C 、T622C、T658C、A750T。关于这9 个碱基突变导致712号酿酒酵母菌株乙醇产量降低的生理机制正在进行深入研究。