鲜食葡萄品种改良岗位
1 材料与方法
1.1 材料
供试材料选取150份葡萄品种(附表1),该群体具有广泛的果粒大小相关性状变异。供试葡萄植株种植于北京市农林科学院林业果树研究所葡萄资源圃(北纬39°58′,东经116°13′)。
1.2 试验方法
1.2.1 样品采集
于2019年4月初萌芽期采取嫩叶,用液氮速冻保存于-80℃冰箱,用于后续DNA提取。
分别于2019—2020年8—10份进行成熟果实样品采集。果实成熟度依据往年物候期记载和果实可溶性固形物含量(ºBrix≥16)确定。由于机械损伤(埋土防寒)、坐果不良或果实腐烂,每年采集的样本数量各不相同。2019年和2020年分别采集到139份和135份葡萄品种果实样品。针对每个葡萄单株分别在阳面和阴面随机挑取3穗果,从不同位置选取10—20个果实作为一个重复,用于后期性状表型调查,3次重复。
1.2.2 葡萄果粒大小相关性状调查
分别对葡萄果实单粒重(BW)、种子数(SN)和种子质量(SW)进行测量分析,测量方法参照刘崇怀等[14]编著的《葡萄种质资源描述规范和数据标准》。
1.2.3 葡萄叶片DNA提取和全基因组重测序
葡萄叶片DNA提取采用植物DNA提取试剂盒(北京天根生物技术有限公司),并利用琼脂糖凝胶电泳检测DNA的完整性和纯度。检测合格的DNA样品通过Covaris破碎机随机打断成长度为350 bp的片段,经末端修复、加ployA尾、加测序接头、纯化、PCR扩增等步骤完成整个文库制备。构建好的文库通过Illumina HiSeqPE150进行测序。
针对原始测序数据,进行过滤,过滤的过程如下:(1)去除接头(Adapter)序列;(2)去除含N比例大于10%的reads;(3)去除低质量reads(质量值Q≤20的碱基数占整条read的50%以上)。进一步利用比对软件BWA将数据比对到参考基因组(http://plants.ensembl.org/Vitis_vinifera/Info/Index),利用Picard(Picard: http://sourceforge.net/ projects/picard/)对比对结果进行排序并标记重复序列。使用软件GATK获得变异数据集,检测到的变异进一步过滤获得高质量标记:(1)非二等位位点去除;(2)第二等位基因频率(minor allele frequency,MAF)小于0.05的位点去除;(3)缺失率大于0.5的位点去除;(4)杂合比例大于0.8的位点去除。
1.2.4 全基因组关联分析(GWAS)
使用Admixture软件(v1.3)[15]进行群体结构的推断。使用PopldDecay软件(v3.41)[16]计算两两标记间的LD大小(r2),绘制连锁不平衡的衰减图。使用gemma软件(v0.98.1)[17]进行GWAS分析,采用GWAS常用模型(GLM、GLM(Q)、MLM(K)、MLM(QK))进行计算。Admixture最优K值对应的群体结构矩阵作为相应模型的Q矩阵,gcta软件计算的样品间亲缘关系矩阵作为相应模型的K矩阵。
1.2.5 候选基因筛选(GO、KEGG注释分析)
根据果粒大小相关性状显著关联的SNP标记在葡萄参考基因组中的物理位置,筛选显著SNP位点上下游LD衰减距离区域范围内的候选基因。利用Ensemble Plants数据库(http://plants.ensembl.org/index.html/)及美国国立生物技术信息中心数据库(https://www.ncbi. nlm.nih.gov/)对候选基因进行功能注释和预测。
1.2.6 数据处理与统计分析
应用Excel 2007和SPSS 13.0软件对性状表型数据进行描述性统计分析和数据分布统计,并运用Pearson相关系数进行性状相关性分析,最低显著水平P<0.01。采用SPSS中paired-samples t test进行显著关联标记等位效应分析。
2 结果
2.1 葡萄果粒大小相关性状统计分析
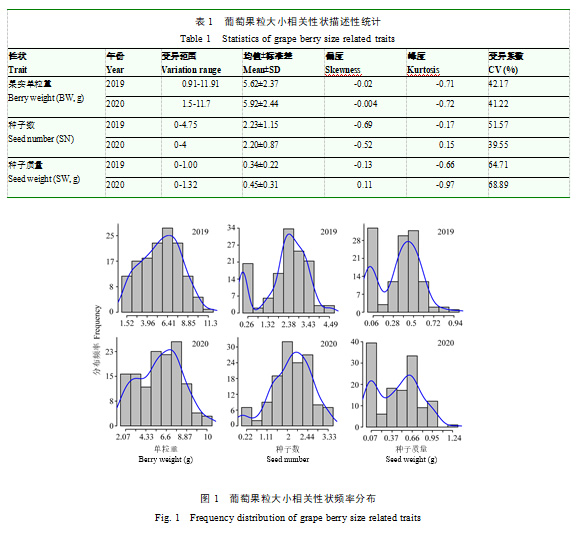
分别于2019年和2020年对150份葡萄品种进行果实单粒重(BW)、种子数量(SN)和种子质量(SW)调查,基本统计分析(表1)表明,2019年关联群体果实单粒重、种子数量和种子质量均值分别为(5.62±2.37)g、(2.23±1.15)个和(0.34±0.22)g;2020年相应各个性状均值分别为(5.92±2.44)g、(2.20±0.87)个和(0.45±0.31)g;不同年份各性状在关联群体中呈现广泛的连续变异,变异系数为39.55%—68.89%。对各个性状的偏度和峰度进行计算,绝对值均小于1,表明这些性状在不同年份均服从正态分布。结合各自的频率分布直方图可以看出(图1),果实单粒重表型数据基本呈现正态分布,种子数量和种子质量呈现非对称性的双峰分布,并不符合常规的单峰正态分布;各个性状均表现数量性状遗传特征。
不同年份关联群体果实单粒重、种子数目和质量的Pearson相关分析表明(表2),果实单粒重与种子数目、种子质量在两个年份均呈显著正相关,种子数目和质量在两个年份呈显著正相关,说明各性状之间相互影响,相互协同。此外,相同性状在不同年份也呈现显著正相关,表明各个性状主要受遗传因素影响。

2.2 全基因组关联分析
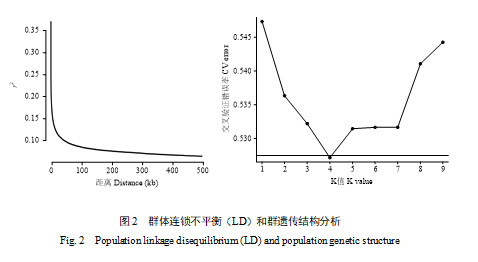
通过软件PopLDdecay以r2降低到全染色体最大值(0.3702)一半时的物理距离作为LD衰减距离(图2-A),计算为1.8 kb。根据连锁不平衡衰减图可见,随着物理距离的增加,r2值急剧下降,表明这些群体在衍化过程中重组率较大,关联分析所需要的SNP密度更大,但相应能获取更小的候选区间,筛选候选基因更加容易。通过Admixture软件分析,当交叉验证错误率最低时,K值为4,因此,可将亚群根据群体的基因型情况细分为4个。将K=4时所对应的群体结构矩阵作为后续关联分析所用矩阵(图2-B)。
连续2年结合果实单粒重、种子数目和种子质量等性状表型数据进行GWAS分析,基于0.01/SNP总数作为显著关联的SNP标记筛选阈值。2019年,共检测到与果粒单重相关的显著SNP有99个,主要位于1号、3号、5号、7号、11号、15号、16号、17号和19号染色体上,表型解释率在14.48%—25.59%(图3-A、附表2);2020年,共检测到与果粒单重相关的显著SNP有73个,主要位于1号、5号、11号、16号和17号染色体,解释表型变异的16.08%— 26.83%(图3-B、附表2)。相较于果粒单重,检测到的与种子数目显著关联的SNP较少,2019年仅检测到1个显著性SNP(图3-C、附表2),表型解释率为24.29%;2020年检测到17个显著性SNP(图3-D、附表2),均位于18号染色体,表型解释率在16.42%—30.48%。两个年份检测到与种子质量显著关联的SNP分别有1个(2019,图3-E)和2个(2020,图3-F),位于18号染色体,解释表型变异的23.59%— 48.29%(附表2)。
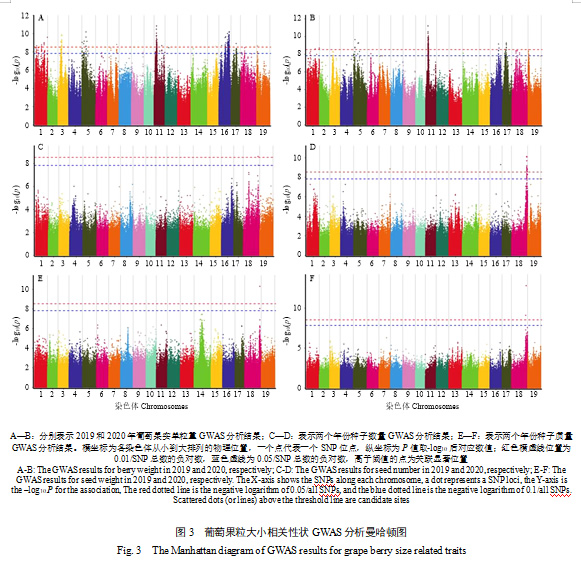
为了保证关联位点的可靠性,进一步筛选两个年份同时检测到的显著SNP位点作为后期候选基因挖掘重点区域,由结果可见(表3、附表2),24个与果粒单重显著关联的SNP在两个年份重复被检测到,分别位于1号、5号、11号和16号染色体上。其中1号染色体有1个SNP位点,2019年和2020年的表型解释率分别为23.45%和23.30%;5号染色体上检测到位于7 537 999和9 220 655位置的两个SNP位点,两个年份表型解释率分别为21.59%、22.03%和22.19%、23.47%;11号染色体上有19个SNP在两个年份均被检测到,表型解释率范围16.03%—27.69%;最后两个SNP位点位于16号染色体,14 893 081位点表型解释率分别为18.70%(2019年)和18.45%(2020年);15 790 424位点2019年和2020年表型解释率分别为18.55%和18.45%。与种子数目关联的显著SNP在两个年份同时检测到1个SNP位点,2019年和2020年表型解释率分别为24.29%和25.67%。而两个年份均检测到的与种子质量显著关联的SNP也是1个,表型解释率达到40.11%(2019年)和48.29%(2020年)。

2.3 候选基因筛选
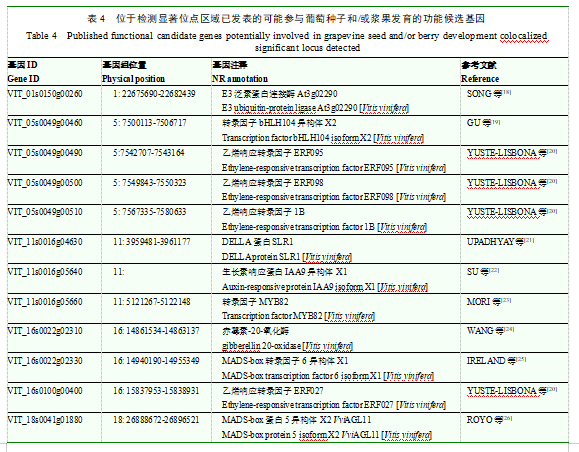
依据该群体的LD衰减距离(图2-A),在26个位点的基因组区段内共检测到76个基因,结合基因功能注释和前人报道,筛选出11个候选基因可能参与果实大小发育过程(表4)。其中与单粒重相关的基因有VIT_01s0150g00260(编码E3泛素蛋白连接酶)、VIT_05s0049g00460(编码转录因子bHLH104)、VIT_05s0049g00490—VIT_05s0049g00510(编码乙烯响应转录因子)、VIT_11s0016g04630(编码DELLA蛋白SLR1)、VIT_11s0016g05640(编码生长素响应蛋白IAA9)、VIT_11s0016g05660(编码转录因子MYB82)、VIT_16s0022g02310(编码赤霉素-20-氧化酶)、VIT_16s0022g02330(编码MADS-box转录因子6)和VIT_16s0100g00400(乙烯响应转录因子ERF027)。与种子数目和质量相关的基因有VIT_ 18s0041g01880(编码MADS-box蛋白VviAGL11)。
2.4 候选基因关联标记遗传效应分析
为进一步确定可能的候选基因,针对与候选基因关联的7个SNP位点的等位变异效应进行分析,6个与单粒重性状显著关联的SNP位点在本群体中呈现3种基因型,与种子数量和质量关联的SNP在本群体中检测到2个基因型。由图4可见,每个SNP的等位变异在两个年份的果实大小相关性状均达到显著水平。例如,SNP 5_7537999位点G/A和A/A等位变异在两个年份的平均果实单粒重显著高于G/G等位变异;SNP 18_26889437不同基因型对种子数目和质量均有显著影响。该结果进一步说明与该标记关联的候选基因可能在调控葡萄果粒大小方面发挥重要的作用。
