育种方法与技术岗位
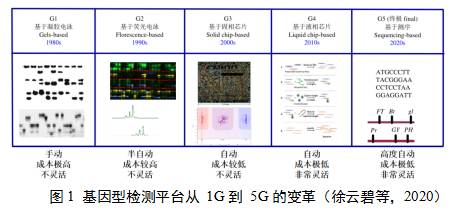
借助于分子标记进行的基因型检测在生物遗传研究与改良等领域发挥着重要的作用。在分子水平进行遗传变异的检测,是分析生物遗传变异的重要手段。对生物体的全基因组进行测序往往要花费巨大的成本,相较而言,以点带面、基于分子标记的遗传变异检测技术则是一种简便、高效、低成本的分子检测手段。基因型检测平台出现至今,随着分子标记的发展以及检测手段的进步,已经经历了多次迭代(图1)。
然而,不同的研究者在他们的研究中往往需要重新开发分子标记。由于使用的分子标记不同,很难直接比较不同的研究结果,造成了巨大的资源浪费。在遗传研究中,迫切需要一种具有差异性、通用性和经济性的分子标记方案来解决目前存在的问题。
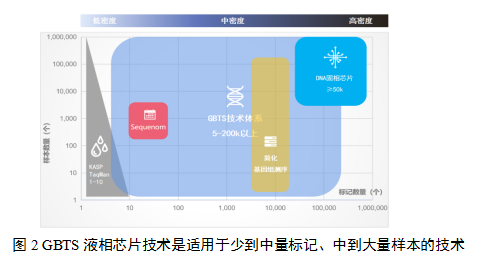
近年来发展起来的目标测序基因分型(GBTS,Genotyping by target sequencing )为解决这一问题提供了新的思路。该技术具有效率高、适用性广、经济等优点。它可以一次检测大量设计的靶点,并对覆盖整个基因组的数千个已知SNP位点进行测序,从而以较低的成本获得全面的基因组信息。而由于其体系中包含了所有目标靶点的引物及其相关的液相捕获体系,因此又被成为液相芯片(Liquid chip)。GBTS技术具有可检测样本数量范围广泛,可检测标记数量范围广泛的双重优点(图2)。与固相芯片相比,GBTS检测更加灵活、样本与标记范围广泛,且成本更低;与简化基因组测序相比,由于标记固定统一,便于不同平台数据的比较。相较于KASP标记,GBTS具有明显的标记数量检测优势。
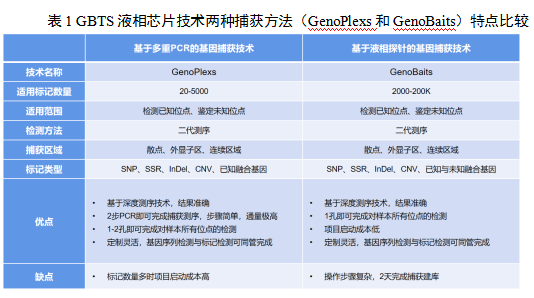
GBTS检测技术按照原理可以分为两类,一类是基于多重PCR技术的GenoPlexs技术,一类是基于液相探针杂交的GenoBaits技术。其具体差异如表1所示。
GenoPlexs的工作原理是依靠PCR对于靶向位点的定点扩增。对多个待测SNP位点设计特异扩增引物,在第一轮PCR中抑制非特异扩增,使数以千计的靶向引物能够在一管PCR反应中实现高度均一化的扩增,从而大量富集目标片段。随后,在第二轮PCR中,加上测序接头和文库条形码,最终获得测序所需的文库。最后通过大规模并行测序(Massively parallel sequencing,MPS)揭示目标位点的标记基因型。目前,该技术已可以一次性满足20-5000个标记的检测。
而GenoBaits工作原理是基于目标探针与靶向序列互补结合进行定点捕获。首先对要测试的材料进行gDNA文库构建。同时根据DNA互补原理,在每个待测位点设计覆盖目标SNP的探针,采用生物素(Biotin)标记对目标探针进行标记。然后,在液态中利用生物素标记的目标探针与基因组目标区域杂交形成双链。随后利用链霉亲和素包衣的磁珠对携有生物素标记的目标探针进行分子吸附,从而捕获与探针杂交的靶点。最后,对捕获的靶点序列进行洗脱、靶点扩增、建库和测序,最终获得目标SNP的基因型。利用GenoBaits技术,目前可以对多达20万个目标位点进行单管同时检测。
目前,GBTS液相芯片技术已经在多种作物的遗传研究中应用。在粮食作物中,GBTS技术已在玉米、水稻、大豆等多种作物中成功应用。在园艺作物中,黄瓜、苹果、西兰花等作物也有GBTS技术应用的报道。而且GBTS技术在许多方面具有广阔的应用前景。其具体的应用范围包括遗传资源评价、生物类群划分、后代鉴定筛选、遗传图谱构建、目的基因定位克隆、性状关联分析等。因此我们认为,GBTS液相芯片技术将成为未来很长一段时间基因型检测的首选,我们也已经成功构建了一款葡萄液相芯片。