华东华南区栽培岗位
徐献斌 耿晓月 李慧 孙丽娟 郑焕 陶建敏
摘要:【目的】为了分析参与调控ABA促进葡萄着色的相关基因,探讨ABA促进葡萄果实花青苷积累的分子机制。【方法】本研究以‘红巴拉多’葡萄为试材,在转色前期(约花后6周)使用300mg·L-1 ABA对果穗进行浸果处理,以清水处理为对照。观察表型并利用超高液相色谱质谱联用仪(UPLC-MS)测定花青苷,再利用转录组测序技术从分子水平对ABA促进花青苷积累的机制进行生物信息学分析。【结果】外源ABA处理3天后葡萄果实着色明显加深,花青苷种类和含量增多。分析ABA处理18小时和3天后葡萄果实转录水平的差异,通过KEGG富集分析发现了52个花青苷生物合成和转运相关基因以及11个ABA信号通路基因。通过分析表达模式,筛选到可能调控花青苷生物合成的转录因子,包括MYB、bZIP、NAC等转录因子。启动子顺式作用元件分析表明,大部分筛选到的差异基因的启动子中含有ABRE元件。对部分候选基因的表达模式进行实时荧光定量(qRT-PCR)分析,证实了RNA-seq的准确性。本研究基于全基因组水平分析ABA处理葡萄果实后的转录组变化,以及花青苷积累相关基因的动态变化和生理反应的分子机制,为揭示ABA促进葡萄花青苷积累的分子机制提供了一定基础。【结论】ABA促进葡萄花青苷积累涉及多个ABA信号转导以及花青苷合成和相关基因,15个转录因子可能参与调控了这一生物过程。
关键词:葡萄;花青苷;ABA;转录组;启动子
0 引言
【研究意义】葡萄(Vitis L.)是一种全球广泛栽培的水果作物,可用于鲜食、酿酒、加工、制汁等,其中鲜食葡萄占有较大比重(约30%)。在我国南方地区避雨栽培模式下,一些红色或黑色品种的葡萄果实,常常因为温度高或光照不足难以着色。果实颜色是衡量鲜食葡萄商业价值的重要指标,成熟期的果实着色不佳严重影响了葡萄的经济价值。在许多鲜食葡萄品种中,外源使用ABA能有效促进葡萄果实着色得到证实,但其潜在的分子机理尚不清楚。因此,探究ABA促进葡萄果实着色分子机制,为生产上外源使用ABA提供理论基础具有重要意义。【前人研究进展】前人研究表明,ABA是调控非呼吸跃变型果实成熟的重要激素,内源ABA含量在果实始熟期开始快速上升,在果实糖分积累、软化、着色等过程中起着关键作用。研究表明,外源ABA处理使得葡萄提早成熟,显著促进葡萄的着色,且不会明显影响可溶性固形物、可滴定酸等果实品质。也有学者认为,外源ABA不仅可以促进葡萄果实着色,同时可以提高葡萄的内在品质。ABA对葡萄果实品质产生不同影响的原因可能是外源ABA的浓度、施用方式以及葡萄品种的不同。但可以证实的是,ABA能促进葡萄果实着色,提高果实的外观品质。在生理水平上,研究者们对外源ABA促进葡萄着色的机理开展了一些研究。外源ABA处理后,果实内源ABA含量提高,内源乙烯合成增加,改变了ABA、乙烯、吲哚乙酸、赤霉素、玉米素核苷等之间的动态平衡关系,从而促进果实花青苷合成和果实着色[9]。在分子水平上,有一些研究从基因角度方面探究ABA促进葡萄着色的机理。外源ABA通过上调VvCHI、VvF3H、VvDFR、VvUFGT、VvF3’5’H、VvOMT、VvGST等类黄酮通路结构基因和VvMYBA1、VvMYBA2、VvMYB5b等转录因子的表达水平,促进花青苷的生物合成。GAO等证实了ABA受体基因VlPYL1在‘巨峰’葡萄中瞬时过表达可以促进花青苷积累。【本研究切入点】尽管前人对ABA促进葡萄果皮花青苷积累方面进行了大量研究,但ABA促进花青苷合成是一个涉及许多基因的复杂过程,其相关的分子机制仍不明确。通过传统分子生物学方法逐一地对葡萄果实花青苷生物合成过程涉及的大量基因进行研究,效率低,难度大。利用高通量转录组测序(RNA-Seq)能够全面、快速地分析ABA促进花青苷合成相关的基因网络变化。【拟解决的关键问题】本研究以‘红巴拉多’葡萄为试材,在转色前期使用ABA处理,选取关键时期进行转录组测序。深入研究与花青苷合成和转运相关基因在ABA处理后的表达模式,并且筛选可能参与调控ABA促进花青苷积累的关键转录因子,以期全面地了解ABA促进葡萄花青苷合成的基因调控网络和表达模式,为ABA促进花青苷积累的分子机制研究奠定基础,同时为生产上外源使用ABA促进葡萄着色提供理论依据。
1 材料与方法
1.1 试验材料
试验于2019年在南京农业大学汤山葡萄基地进行。葡萄是典型的非呼吸跃变型果实,而红色葡萄品种在果实发育过程中伴有花青苷合成,是研究ABA促进花青苷生物合成的良好试材。试验选用‘红巴拉多’葡萄为试验材料,选择树体长势一致的植株,树形为平棚架型,栽培管理等同常规。
在果实的转色前期(花后第六周),在预实验基础上选用300 mg·L-1 ABA(上海源叶)对‘红巴拉多’葡萄果穗进行浸果处理,以清水处理为对照。处理后24h内每隔6h取样一次,处理后3d(出现明显表型差异)取样一次。将所取样品用手术刀片剥取果皮,液氮速冻后-80℃保存备用。
1.2 花青苷的提取及定性分析
总花青苷的提取参照Jia等[13]的方法并加以改进:取待测定的葡萄果皮样品,用液氮研磨成粉末,称取1.0g样品粉末,加入10mL含1%盐酸的甲醇溶液,4℃黑暗条件下静置24h,10000rpm高速离心10min,沉淀反复浸提两次,合并上清液,用0.22μm滤膜过滤后合并上清液待测。
使用AB SCIEX Triple TOF 5600+液质联用仪测定花青苷的组分及相对含量,参照Zhang等[14]的方法并加以改进:流动相A泵溶液:0.1%甲酸水溶液,B泵溶液:0.1%乙腈。洗脱条件:流动相洗脱梯度:0-0.5min,5% B;0.5-20.5 min,5-40% B;0.5-22.5min,40-95% B。流速:0.2 mL·min-1。进样量:2μL。
质谱条件:使用正离子模式下的电喷雾源和质谱采集模式,选择的质量范围为50–1200 m/z。使用亮氨酸脑啡肽(m/z 556.2771)重新校准,启用了锁定质量选项。电离参数为:毛细管电压3.0kV,锥电压40V,源温度120℃、脱溶气体温度为400℃。
采用外标法对花青苷组分进行相对定量:配制0.02-20ng·mL-1不同浓度的锦葵色素-3-O-葡萄糖苷标准品溶液绘制标准曲线,相关系数为0.9986,回归方程为y=1E+06x-245385。
1.3 ABA含量测定
ABA的提取和测定参照Hu等[15]的方法并加以改进:取待测定的葡萄果皮样品,用液氮研磨成粉末,称取1.0g样品粉末,加入10mL含80%的甲醇溶液,4℃黑暗条件下静置12h,10000rpm高速离心10min,沉淀反复浸提两次,合并上清液,加入0.2g PVPP,在4℃恒温摇床中120rpm震荡1h,离心后取上清液过C18小柱,使用冷冻干燥器冻干48h,再加入1mL 80%的甲醇溶解,用0.45 μm滤膜过滤后合并上清液待测。
HPLC条件:所用仪器为3200 Qtrap 高效液相色谱-串联三重四级杆质谱联用仪(AB SCIEX),色谱柱:phenomenex Kinetex® XB-C18 (100 * 3 mm, 2.6 μm); 流动相:A泵溶液:0.1% 甲酸水溶液,B泵溶液:甲醇。洗脱条件:0 -1 min,5% B;1 - 4 min,5% B-95% B;4 - 8 min,95% B;8 - 8.1 min,95% B -5% B。流速:0.3 mL· min-1;柱温:40 ℃;进样量:10μL。
质谱条件:在MRM模式下,用电喷雾电离ESI负离子模式,离子源:Turbo Spray,气帘气CUR:25,离子源喷雾电压IS:-4500 kV, 温度TEM: 500℃,喷雾气GS1: 45, 辅助加热气GS2: 30, 碰撞气CAD:Medium, 入口电压EP: -10V, 碰撞单元出口电位CEP: -18.37, 出口电压CXP: -2.2V。
精确配制5-1000ng·mL-1不同浓度的ABA标准品溶液绘制标准曲线,相关系数为0.99913,回归方程为y=127.22x+145.81。
1.4 总RNA提取、cDNA文库建立及测序
总RNA的提取采用多糖多酚植物总RNA提取试剂盒(TIANGEN, China),所有操作按照说明书进行。取2μL RNA样品进行琼脂糖凝胶电泳,检测RNA样品的质量。用Nanodrop ND-1000 Spectrophotometer 检测RNA浓度。
使用NEBNext® Ultra™ RNA 文库制备试剂盒(NEB,USA)建立测序文库。使用Qubit2.0荧光仪和Agilent 2100 bioanalyzer生物分析仪检查cDNA文库质量。检测合格的cDNA文库在Illumina Hiseq平台上测序。每个样品进行三次生物学重复。将原始数据进行过滤,去除衔接子序列、包含poly-N和低质量reads后,获得clean reads。
1.5 葡萄参考基因组比对及基因功能注释
利用HISAT2将clean reads与葡萄参考基因组(ftp://ftp.ncbi.nlm.nih.gov/genomes/all/GCF/000/003/745/GCF_000003745.3_12X/GCF_000201 003745.3_12X_genomic.fna.gz)进行比对。计算每千个碱基的转录每百万映射读取的fragments(FPKM值),用RSEM工具检测基因和转录表达水平,用DEseq2检测差异表达基因(DEGs)。筛选差异基因的错误发现率(FDR)阈值设为≤0.05。差异基因基于NCBI非冗余蛋白序列数据库(Nr ,http://ftpprivate.ncbi.nlm.nih.gov) Gene Ontology数据库(GO,http://www.geneology.org/) 、KEGG Ortholog数据(KEGG, http://www.genome.jp/kegg/)进行基因功能注释。
1.6 差异表达基因富集分析
根据Kyoto Encyclopedia of Genes and Genomes(KEGG)的注释结果和官方分类,分别对差异基因的功能和生物学途径进行分类。用Blast2Go进行GO和富集分析, p-value(Padj)≤0.05。采用两种统计检验(hypergeometric Fisher's exact test, q<0.01)进行统计分析,以确定KEGG途径在统计学上的显著富集。
1.7 转录因子(TF)分析及启动子顺式作用元件分析
将差异基因与葡萄转录因子数据库(http://planttfdb.cbi.pku.edu.cn/index.php?sp=Vvi)进行比对,获取差异表达的转录因子以进行进一步筛选。
利用TBtools(http://www.tbtools.com/)软件和葡萄全基因组序列提取候选差异基因上游2kb启动子序列,上传到plantcare(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)网站进行顺式作用元件分析。
1.8 qRT-PCR分析
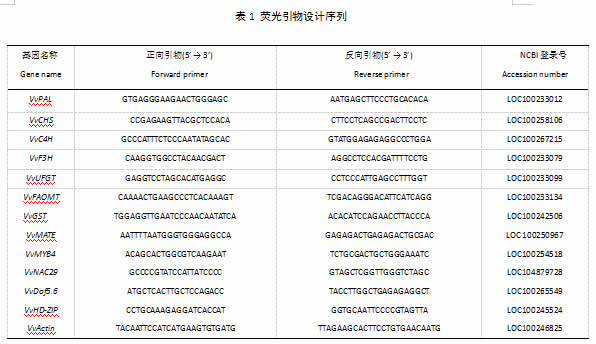
qRT-PCR引物使用Beacon Designer 7.0软件(Premier Biosoft Internaition,USA)设计,引物见表1。所有引物均用PCR扩增、电泳和溶解曲线进行测试以保证引物特异性。使用ABI-7300系统进行qRT-PCR,反应体系按SYBR Green PCR Master Mix (Takara)试剂盒说明书进行。以VvActin为内参,用2-△△Ct公式计算相对表达量。
2 结果与分析
2.1 ABA处理后‘红巴拉多’葡萄果皮花青苷含量和组分分析

如图1所示,‘红巴拉多’葡萄在外源ABA处理3天后外观出现了明显的变化,果皮呈现红色。UPLC-MS结果显示:与对照组相比,ABA处理显著提高了葡萄的总花青苷含量,ABA处理组葡萄果皮的总花青苷含量为对照的9倍左右。对照组葡萄果皮中只有飞燕草素 3-O-(6''-p-香豆酰葡萄糖苷)、芍药素 3-O-葡萄糖苷、锦葵色素 3-O-葡萄糖苷三种花青苷,且含量很低。与对照组相比,ABA处理组增加了三种花青苷,分别是矮牵牛素 3-O-葡萄糖苷、天竺葵素 3-O-(6''-p-香豆酰葡萄糖苷)、芍药素 3-O-(6''-p-香豆酰葡萄糖苷),但含量并不高。值得注意的是,ABA处理显著增加了芍药素 3-O-葡萄糖苷、锦葵色素 3-O-葡萄糖苷的含量,ABA处理组的含量分别约为对照组的12倍和9倍。

2.2 VvNCED1基因表达模式分析及内源ABA含量测定
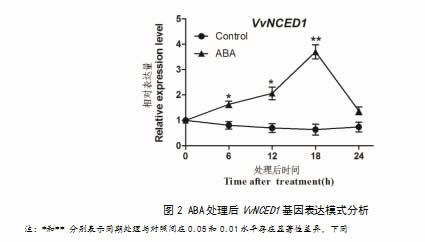
qRT-PCR结果显示,ABA处理后24h内 VvNECD1的表达随着处理时间表现出先升后降的变化趋势,在处理后18h达到峰值,随后逐渐下降。ABA处理后VvNECD1相对表达水平显著高于对照,说明葡萄果皮中VvNCED1的转录受到了ABA的迅速诱导而上调。
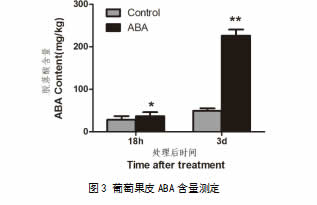
对葡萄果皮ABA含量进行HPLC-MS分析,结果表明ABA处理显著提高了果皮中ABA含量。在处理后18h,ABA处理组果皮ABA含量约为对照组的1.7倍,而在处理后3d天则为6.2倍。
2.3 转录组数据分析
利用 Illumina HiSeq 高通量测序平台对已构建好的12个 cDNA 文库进行测序,得到4135.2—4852.8万条不同数目的原始序列。数据过滤后得到4005.4—4733.7万条有效序列,有效比例96.19%—97.87%。与葡萄参考基因组比对后得到3578.7万—4225.3万条匹配的序列,比对率为94.3%—95.33%。通常质量控制参数 Q30>80%表示转录组测序质量可靠,本试验中 Q30> 94.3%表明测序数据质量很高,可用于ABA处理后葡萄果皮差异表达基因的挖掘及对ABA促进葡萄果实着色机制的探讨。
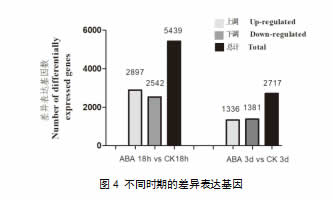
将处理后18h和处理后3d两个时期的ABA处理组和对照组转录组数据进行比对分析,分别获得5439和2717个差异表达基因。在处理后18h,共计2897个基因表达上调,2542个基因表达下调;在处理后3d,共计1336个基因表达上调,1381个基因表达下调。

2.4 KEGG富集分析
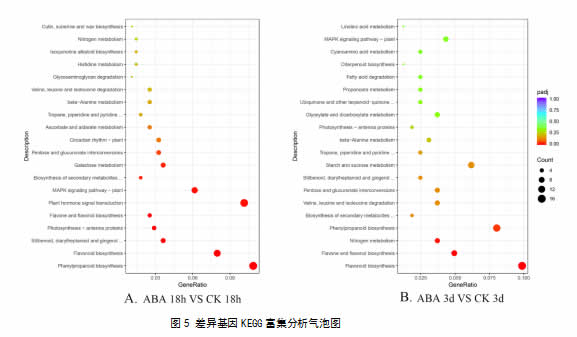
为了进一步分析预测差异基因的生物功能,对ABA 18h vs CK 18h和ABA 3d vs CK 3d两组差异基因进行了KEGG富集分析,分别有2529个差异基因富集到118个通路、1335个DEGs富集到113个通路上。如图5所示,黄酮和黄酮醇生物合成、类黄酮生物合成以及植物信号转导在两组差异基因的KEGG分析中显著富集。
2.5 ABA信号通路差异表达基因分析
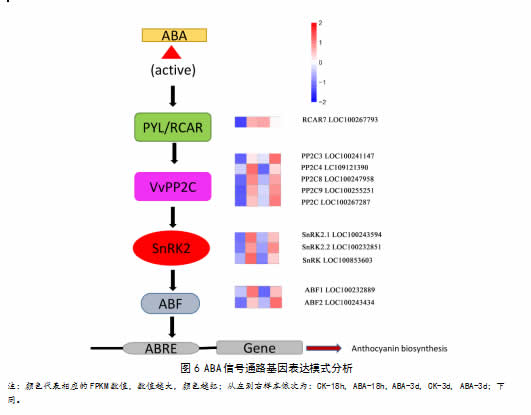
差异表达基因在 KEGG Pathway 数据库中注释分析,获得植物信号转导途径(KEGGID:vvi04075)中ABA信号通路的差异基因。11个差异表达基因中有1个基因被注释为ABA受体(PYL/PYR),5个被注释为2C型蛋白磷酸酶(PP2C)、3个基因被注释为SnRK2激酶、2个基因被注释为ABRE元件结合因子(AREB/ABFs)。通过绘制热图对差异表达基因在两个时期的表达水平进一步分析(图6),结果显示:ABA受体VvRCAR7表达水平在ABA处理后18h上调,在ABA处理后3d下调。除VvRCAR7外的其他10个ABA信号通路基因表现出相似的表达模式,与对照相比,差异基因的表达水平在ABA处理后18h和ABA处理后3d均上调。与ABA处理3d后相比,在ABA处理18h后VvPP2C4、VvPP2C8、VvSnRK2.1、VvSnRK、VvABF1表达水平较高,VvPP2C3、VvPP2C9、VvPP2C、VvSnRK2.2、VvABF2表达水平较低。
2.6 花青苷合成和转运相关基因的表达
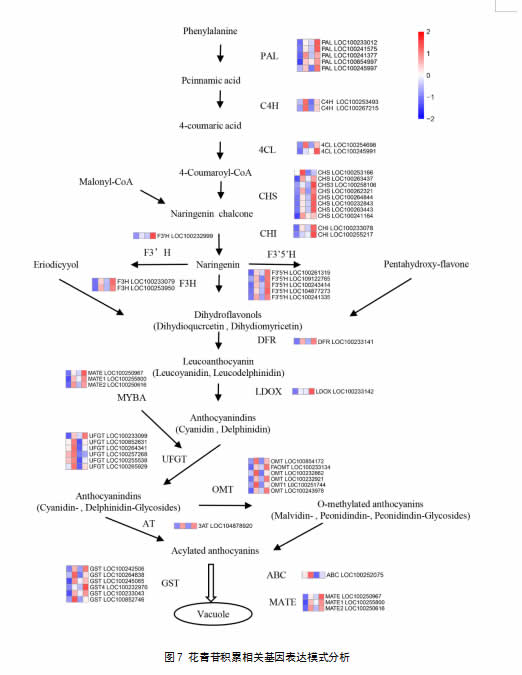
差异表达基因在 KEGG Pathway 数据库中注释分析,获得类黄酮生物合成途径(KEGGID: vvi00941)的差异基因。5个基因被注释为苯丙氨酸氨基裂解酶 (Phenylalanine ammonia-lyase, PAL),2个基因被注释为肉桂酸4-羟化酶(cinnamate-4-hydroxylase,C4H),2个基因被注释为4-香豆酰CoA连接酶(4-coumaroyl-CoA synthase,4CL),8个基因被注释为查尔酮合酶(chalcone synthase,CHS),2个基因被注释为查尔酮异构酶(chalcone isomerase,CHI),1个基因被注释为类黄酮3′-羟化酶(flavonoid-3′-hydroxylase ,F3′H), 5个基因被注释为F3′5′H,2个基因被注释为黄烷酮3-羟化酶(flavanone-3β-hydroxylase,F3H),1个基因被注释为黄烷酮醇4-还原酶(dihydroflavonol-4-reductase,DFR),1个基因被注释为无色花色素双加氧酶,6个基因被注释为UDP葡萄糖-类黄酮3-O-葡萄糖基转移酶(flavonoid glucosyltransferase,UFGT)。
根据基因功能注释,在差异基因列表中查找与花青苷修饰和转运相关的差异表达基因。差异基因中有6个基因与花青苷修饰有关,5个被注释为O-甲基转移酶 (O-methyltransferase, OMT)和1个花青苷酰基转移酶(anthocyanin acyltransferases,AAT)。此外,有11个差异基因与花青苷转运相关,其中有6个被注释为谷胱甘肽转移酶(glutathione S-transferase,GST)、1个被注释为ATP 结合盒亚族C (ATP binding cassette C family,ABCC)、4个被注释为多药和有毒化合物排出家族(multidrug and toxic compound extrusion,MATE)。
对花青苷生物合成结构基因、修饰和转运相关基因的表达模式进行分析(图7),52个差异基因均在ABA处理后表达水平上调。与ABA处理18h后相比,18个差异基因在ABA处理3d后表达水平较低,34个差异基因表达水平较高。值得注意的是,2个C4H基因均在ABA处理18h后表达水平最高,5个UFGT基因中有4个也在ABA处理18h后表达水平最高。
2.7 花青苷生物合成相关转录因子的筛选
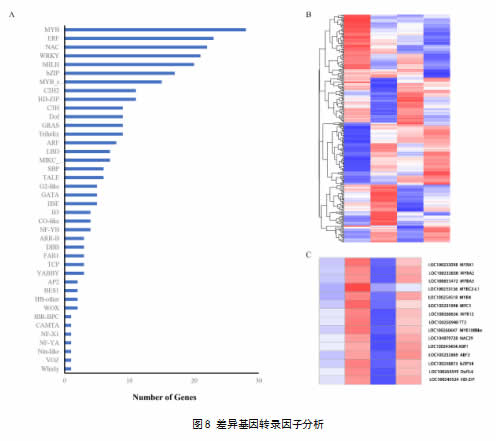
基于转录组测序数据中差异表达的转录因子,参考北京大学植物转录因子数据库PlantTFDB(http//planttfdb.cbi.pku.edu.cn/)公布的葡萄转录因子家族序列信息,共获得 296 个差异转录因子。如图8所示,对差异表达转录因子进行划分,共分为40个转录因子家族。含有转录因子最多的三个家族是MYB家族、ERF家族、NAC家族,分别含有28、23、23个转录因子。其他含有转录因子较多的是WRKY、bHLH、bZIP家族,分别有21、20、17个转录因子。
对所有差异表达的转录因子进行表达模式分析(图8),并筛选与VvMYBA1表达模式相似的转录因子。共得到了14个可能与花青苷生物合成相关的转录因子,主要有MYB家族的转录因子。也有少量的bZIP、bHLH、NAC、Dof、HD-ZIP转录因子。这些转录因子涉及花青苷生物合成、ABA信号转导、抗逆胁迫、果实成熟等多个生物代谢途径。
2.8 差异基因启动子序列ABRE元件统计
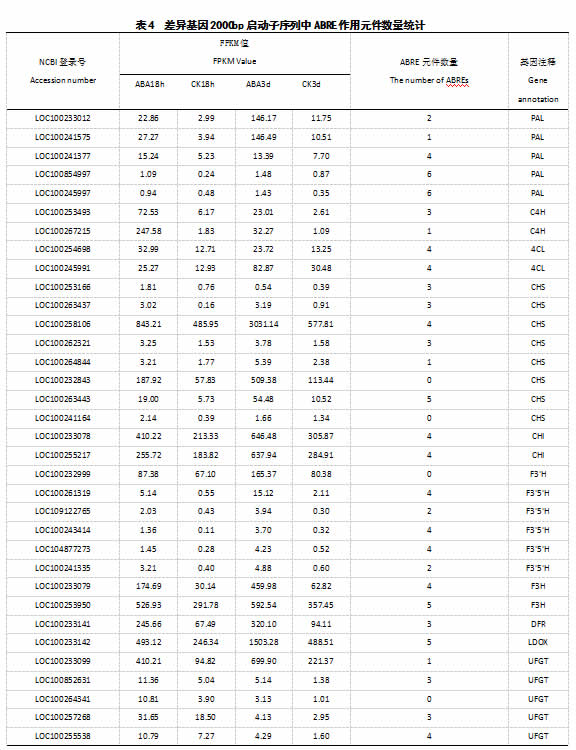
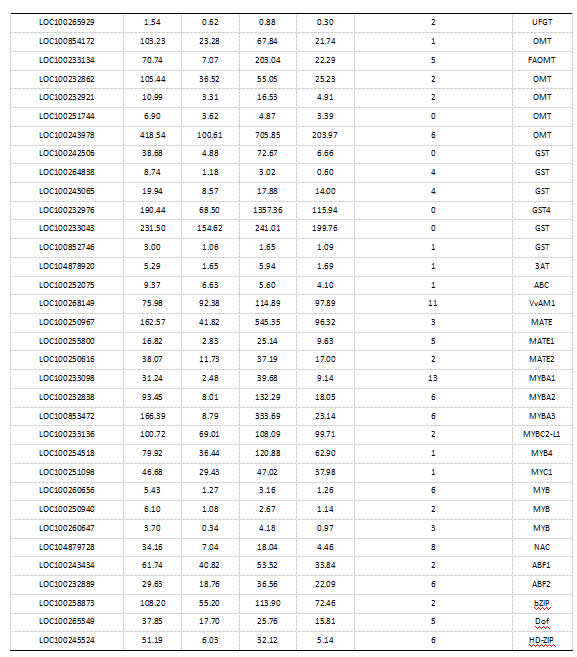
候选差异基因启动子中ABA响应元件(ABRE)数量统计结果显示(表4):60个差异基因的启动子序列中含有ABRE元件,有50个启动子中含有多个ABRE元件。类黄酮生物合成途径中的32个差异基因启动子序列中含有ABRE元件,只有F3'H(LOC100232999)、CHS(LOC100232843 、LOC100241164)、UFGT(LOC100264341) 4个基因的启动子序列中不含有ABRE元件。与花青苷修饰相关的7个差异基因中,只有OMT (LOC100251744)的启动子序列不含有ABRE元件。与花青苷转运相关的11个差异基因中,只有3个GST(LOC100242506、LOC100232976、LOC100233043)的启动子序列中不含有ABRE元件。15个可能参与调控花青苷生物合成的转录因子的启动子序列中均含有ABRE元件。值得注意的是,与花青苷转运相关的VvAM1启动子中含有11个ABRE元件,调控花青苷生物合成的转录因子VvMYBA1启动子中含有13个ABRE元件。
2.9 差异表达基因qRT-PCR验证
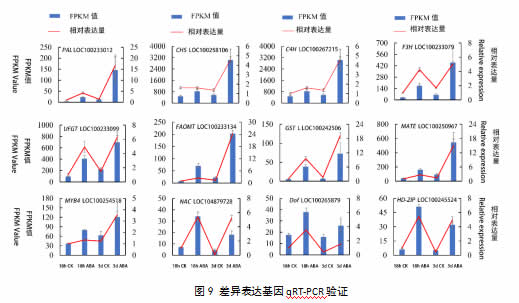
选取6个花青苷生物合成相关基因、2个花青苷转运相关基因和4个差异表达转录因子进行qRT-PCR分析。如图7所示,12个差异表达基因均在ABA处理后表达上调,其中C4H、NAC、Dof、HD-ZIP处理后18h相对表达水平最高,PAL、CHS、F3H、UFGT、FAOMT、GST、MATE、MYB4均在处理后3d相对表达水平最高。以上qRT-PCR结果均与转录组测序结果相符,说明转录组测序数据准确可靠。
3 讨论
前人研究表明,花青苷的含量和组分决定了葡萄着色的类型。外源ABA能促进葡萄果皮中总花青苷和花青苷单体的含量。本研究中,外源ABA处理3d后‘红巴拉多’葡萄葡萄果实明显呈现红色,而清水处理的葡萄仍为绿色(图1)。通过UPLC-MS测定花青苷发现,外源ABA处理增加了葡萄果皮花青苷的种类和含量。ABA处理组增加的矮牵牛素 3-O-葡萄糖苷、天竺葵素 3-O-(6''-p-香豆酰葡萄糖苷)、芍药素 3-O-(6''-p-香豆酰葡萄糖苷)含量很低,花青苷种类的增加可能不是葡萄迅速着色的原因。外源ABA处理后,葡萄果皮中芍药素 3-O-葡萄糖苷、锦葵色素 3-O-葡萄糖苷两种单体花青苷含量的显著提高可能促进了葡萄着色。
ABA是调控葡萄成熟的关键内源激素,其含量的变化与果实成熟进程高度相关。9-顺式-环氧类胡萝卜素双加氧酶(NCED)是植物体内ABA生物合成的关键限速酶。VvNCED1在在葡萄果实始熟期启动ABA的合成,在果实成熟过程中起着关键调控作用。曾有研究表明,外源ABA处理葡萄果实后VvNCED1表达迅速上调,同时内源ABA含量增加。本试验qRT-PCR结果也显示,外源ABA处理显著上调了VvNCED1的表达且在处理后18h达到峰值。HPLC-MS分析发现,ABA处理后18h和处理后3d,内源ABA含量均显著增加。外源ABA可能通过激活VvNCED1的转录,启动了葡萄果实中内源ABA的合成,从而促进了葡萄果实的花青苷积累和着色。
ABA信号通路由ABA受体(PYL/RCAR)、2C型蛋白磷酸酶(PP2C)、SnRK2激酶、ABRE元件结合因子(AREB/ABFs)组成。葡萄基因组中鉴定和分离出了7个ABA受体基因VvPYLs,这些基因受不同非生物胁迫因子的调控。研究表明VlPYL1参与调控葡萄果实花青苷的生物合成。本实验中VvPYL1在ABA处理后18h表达上调,说明VvPYL1可能在ABA诱导的花青苷积累中发挥调控作用,这与前人研究一致。葡萄中有两个AREB/ABFs 类基因,被命名为VvABF1和VvABF2。其在荔枝中的同源基因LcABFs也被证明参与调控荔枝果皮花青苷生物合成过程。转录组数据显示,VvABF1和VvABF2在ABA处理后两个时期的转录水平均上调,可能在调控花青苷生物合成方面与LcABFs作用相似。共有11个ABA信号通路基因在ABA处理后表达上调,说明葡萄果实ABA信号转导在花青苷合成中可能具有重要作用。
葡萄果皮中花青苷的生物合成主要通过类黄酮通路进行,这个过程由结构基因所控制,调节下游各个结构酶形成多酶复合物,进而催化花青苷生物合成。本研究有35个差异基因富集到类黄酮通路中,包括了PAL、C4H、4CL、CHS、CHI、F3′H、 F3′5′H、F3H、DFR、LDOX、UFGT等11个结构基因。ABA处理后这些结构基因表达上调,类黄酮通路合成增加,催化了花青苷的合成。O-甲基转移酶 (OMT)和花青苷酰基转移酶(AAT)可将花青素进行修饰,使其更稳定和多样化。差异基因中的5个OMT和1个AAT也在ABA处理后表达水平增加,表明ABA处理后花青苷修饰过程活跃。谷胱甘肽转移酶(GST)、ATP 结合盒亚族C (ABCC)、多药和有毒化合物排出家族(MATE)是将花青苷从细胞质转移到液泡中的关键基因。11个花青苷转运相关基因上调,说明ABA处理后花青苷转运过程增强。本研究发现,ABA对葡萄花青苷的影响是一个复杂的过程,涉及合成、修饰、转运等多个过程。
花青苷生物合成涉及许多转录因子的调控,VvMYBA1是调控葡萄花青苷生物合成重要的转录因子。VvMYBA1在自身启动子的作用下转入白皮品种‘霞多丽’葡萄植株,转基因葡萄果皮变红,在35S启动子的作用下过表达,转基因植株的果皮果肉均积累花青苷呈现黑紫色;而VvMYBA1在黑皮品种‘西拉’葡萄中沉默后,转基因植株果皮花青苷积累很少或不积累,呈现红皮或者白皮的表型[32]。有研究表明 VvMYBA1通过激活 VvUFGT、VvDFR 的启动子来调节其表达,从而调控花色苷的合成积累。功能相似或参与同一生物合成过程的转录因子往往表现出相似的表达模式,因此与VvMYBA1表达模式相近的转录因子可能与花青苷生物合成有关。本研究筛选得到的15个转录因子中,6个转录因子已被证明参与调控花青苷生物合成。VvMYBA1、VvMYBA2和VvMYBA3在葡萄2号染色体上形成基因簇,VvMYBA1和VvMYBA2决定了葡萄是否着色以及着色类型。有研究表明MYB家族成员VvMYB12可能正调控类黄酮的合成。VvMYC1属于bHLH类转录因子,研究证实VvMYC1与MYB5a、MYB5b、MYBA1、MYBA2和MYBPA相互作用参与调控类黄酮途径。2个转录抑制因子VvMYBC2L1和VvMYB4-like负调控花青苷生物合成过程。转录抑制因子VvMYBC2L1通过与bHLH结合或抑制VvOMT和VvGST4的表达负调控花青苷的合成。另外一个R2R3转录抑制因子VvMYB4-like在烟草中过表达引起花的色素积累减少,在拟南芥中过表达使下调了花青苷生物合成结构基因AtANS、AtDFR、AtUFGT的表达水平。负调控花青苷生物合成的VvMYBC2L1和VvMYB4-like表达水平在ABA处理后上调,这可能是一种花青苷合成的反馈调节机制。许多研究表明,在花青苷积累过程中R2R3-MYB转录激活子可能会上调R2R3-MYB转录抑制子的表达,从而提供反馈抑制使得花青苷处于适当的水平。此外,还有8个差异表达的转录因子可能参与调控花青苷生物合成,其中包括2个MYB (TT2, LOC10050942; MYB108-like, LOC100260647)、1个NAC(NAC29, LOC104879728)、3个bZIP (ABF1, LOC100243434 ; ABF2, LOC100232889; bZIP34, LOC100258873)、1个Dof (Dof5.6, LOC100265549)、1个HD-ZIP (ATHB-12, LOC100245524)。这些转录因子均在ABA处理后表达上调,在花青苷生物合成过程中的调控作用尚需进一步实验验证。
启动子是控制基因表达的重要转录调节因子,分析启动子序列中的顺式作用元件是了解基因转录调控表达模式及调控机制的关键。ABRE元件是一种含有G-box的保守顺式作用元件,一般存在于ABA所调控基因的启动子中,ABA通过启动子区域的ABRE元件激活下游基因的表达。为了研究ABA对差异表达基因的调控作用,对差异基因启动子进行提取和顺式作用元件分析。60个候选差异基因的启动子中含有ABRE元件,这些启动子可能是ABA诱导型启动子,可在ABA信号的刺激下快速调整转录活性,从而调控基因的转录水平。在先前的多个研究中,VvMYBA1受到ABA的诱导而显著上调。本研究发现VvMYBA1启动子序列中多达13个ABRE元件,说明VvMYBA1可能是ABA直接调控的基因,这与前人研究相符。大多数与花青苷合成、修饰、转运相关的基因以及相关转录因子,启动子序列中含有ABRE元件,说明它们可能是ABA诱导型基因。由此推测,与葡萄花青苷积累相关基因在ABA处理后转录水平上调,可能与启动子中ABRE元件的转录激活效应有关。外源ABA处理后,葡萄果实内源ABA水平提高,ABA信号转导活跃,激活了下游一系列与花青苷积累相关基因的转录,从而使得花青苷合成和转运增强,在果皮中积累而使葡萄着色。
4 结论
ABA促进葡萄花青苷积累涉及多个ABA信号转导、花青苷合成和转运相关基因,15个转录因子可能参与调控了这一生物过程。