种质资源收集与评价岗位
孙磊 樊秀彩 张颖 姜建福 刘崇怀
葡萄果实颜色性状呈现明显的连续变异特点,且在一定程度上受环境因素影响,表现出典型的数量性状特点。虽然对于MYBA基因的研究很好的揭示了葡萄红色系和黄绿色系果实颜色变异的分子机制,但是作为数量性状,红色系葡萄果实着色类型多样,仅这一个位点难以完全解释果实颜色变异的内在调控机理。为了解决此问题,近些年也开展了一些关于葡萄果实颜色的QTL定位研究。但是,这些QTL定位均是基于传统分子标记构建的低密度遗传图谱,主要还是定位到了位于2号染色体MYBA 基因所在的位点,难以准确定位到其他重要的微效QTL位点。本研究利用‘红地球’和‘玫瑰香’的F1代杂交群体,对其进行全基因组重测序,基于开发的丰富SNP标记构建高密度遗传图谱。结合杂交群体果实着色情况,从不同角度对果实颜色性状进行QTL定位,定位到多个与葡萄果实颜色及花色苷积累相关的QTL位点,为葡萄果实颜色调控的进一步研究奠定了基础。
1 材料与方法
1.1 试验材料
实验材料为‘红地球’‘玫瑰香’以及它们的95株杂交后代。
1.2 杂交群体果实着色情况
根据杂交群体成熟期果实的整体着色情况,其果实着色被分为‘0-5’级,‘0’代表绿色和黄绿色着色类型,‘1-5’代表红色系后代果实颜色由浅到深。此外,使用柯尼卡美能达(日本)色差计(CR-410)对杂交群体成熟期果实的着色情况进行调查,每个单株至少选取10粒果实,每粒果实选取5个点进行色差检测。判断果实成熟采用观察种子颜色和检测可溶性固形物相结合的方法,种子完全变褐,可溶性固形物平均达到18以上且连续一周不再升高,便可判断果实处于成熟期。
1.3 基因组DNA提取、文库构建
基因组DNA提取方法详见
2.2.2。经1%琼脂糖凝胶电泳和微量紫外分光光度计检测提取基因组DNA的完整性和浓度,检测合格后进行基因组重测序文库的构建。取1μg基因组DNA,使用超声波破碎仪(Covaris M220)将其随机打断,并使用AxyPrep Mag PCR清洁试剂盒将片段进行纯化。使用末端修复混合液(End Repair Mix)对打断的DNA片段进行末端修复,随后使用平末端PCR产物添A试剂盒(A-Tailing Kit)对修复的DNA片段进行3’端加A。将Illumina测序接头连接到加A的DNA片段3’端,并进一步对连接产物进行纯化。选择合适大小的DNA片段,在PCR仪上对连接产物进行PCR扩增,以提高连接有测序接头的DNA片段的丰度。使用生物芯片分析系统(Agilent Technologies 2100bioanalyzer)和实时荧光定量PCR系统(ABI StepOnePlus Real-timePCR System)对构建的文库进行质量检测。将质量检测合格的文库在Illumina Hiseq X平台上进行PE150测序。亲本‘红地球’和‘玫瑰香’的测序深度选择30X,杂交后代的测序深度选择10X。
1.4 测序数据的过滤及比对
使用Soapnuke软件去除含测序接头的、低质量的以及N碱基的比例大于1%的reads,得到高质量的Clean data数据用于后续的比对分析。使用短序列比对软件 BWA(0.7.15)的“mem”算法(比对参数为:-t 5 -k 30 -M –)将过滤后得到的clean data 比对到葡萄参考基因组上(PN40024 assembly12X)。
1.5 群体SNP的检测与标记的开发
采用GATK(3.7)流程中的分析工具, 对处理后的比对结果文件进行变异检测。首先进行一次初步的变异检测并构建一个可信度较高的已知的SNP和indel数据集,利用该结果对比对结果reads的碱基质量值进行重新校正(BaseRecalibrator和PrintReads工具),使校正后的比对文件中reads的碱基质量值能够更加接近与参考基因组之间真实的错配概率,然后使用校正后比对文件进行后续的变异检测。然后使用变异检测工具HaplotypeCaller获取每个个体每条染色体的gvcf变异检测结果文件。最后利用GenotypeGVCFs工具将个体gvcf进行整合,最终得到群体变异结果,并用SelectVariants和VariantFiltration工具对检测到的变异进行进一步的提取和过滤(过滤参数:QD<2.0/FS>60.0/MQ <40.0/MQRankSum<-12.5/ReadPosRankSum<-8.0),以得到高度可信的群体SNP。对得到的群体SNP进一步进行过滤,过滤掉reads支持数(DP)小于10、质量值(GQ)小于40、缺失率大于20%以及非二态型的SNP位点,以获得高质量的群体SNP标记。
1.6 遗传图谱构建
使用L e p M a p 3 软件进行图谱构建。首先使用该软件的ParentCall2功能筛选得到可用于软件分析的information marker。然后使用该软件的Filtering2功能(参数:dataTolerance=0.1;removeNonInformative=1)对偏分离标记进行过滤,过滤掉子代偏分离的基因型位点。再使用该软件的SeparateChromosomes2功能进行连锁群的划分,将LOD值设置为7时,可将所有标记划分为19个连锁群,且与基因组的染色体的信息对应性较好。然后再使用JoinSingles2All功能将未能划入连锁群的single marker尝试再划入连锁群中。最后使用OrderMarkers2功能计算各个连锁群的遗传距离。最终获得该群体的高密度遗传图谱。
1.7 QTL定位
利用构建的遗传图谱,结合第二章的群体果实颜色的表型数据,使用MapQTL 6.0软件进行QTL定位。选择MQM mapping分析方法,使用的算法为Kosambi’s。
2 结果与分析
2.1 杂交群体果实着色情况调查
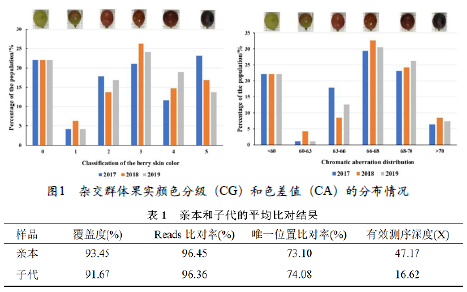
通过2017、2018和2019三年的果实颜色性状调查,发现红地球和玫瑰香杂交群体的果实颜色从黄绿色到紫黑色具有明显的性状分离(图1)。通过果实颜色分级,将群体果实着色情况分为6级,‘0’表示黄绿色果实株系,‘1-5’表示杂交后代的果实颜色从粉红色到紫黑色逐渐加深。遗传群体中,21株黄绿色果实类型的杂交后代非常稳定,每年果实颜色均为黄绿色。在红色果实株系中,果实颜色为‘3’级的占比最多,占到了群体的21%(2017)到26%(2018),群体果实颜色从‘1’级到‘5’级基本呈正态分布特征(图1)。
2.2 杂交群体基因组重测序数据统计
对亲本和杂交后代进行基因组重测序之后,将原始数据中含测序接头的、低质量的以及N碱基的比例大于1%的reads进行过滤,在两个亲本和95株杂交后代中分别获得42.72 Gb和715.24 Gb高质量的Clean data。亲本Clean reads的Q20和Q30平均准确率分别为98.57%和96.49%,子代Clean reads的Q20和Q30平均准确率分别为98.49%和96.27%。亲本Clean reads的平均GC含量为36.72%,子代为37.13%。
2.3 序列比对及群体SNP标记的开发
将Clean reads与参考基因组进行比对,亲本和子代的平均覆盖度分别为93.45%和91.67%,平均Reads比对率分别为96.45%和96.36%,平均Reads基因组唯一位置比对率分别为73.10%和74.08%,平均有效测序深度分别达47.17 X和16.62 X(表1)。通过过滤,获得27454个SNP标记用于高密度遗传图谱的构建。
2.4 高密度遗传图谱的构建
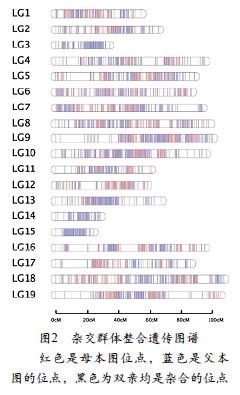
利用开发的SNP标记分别构建了杂交群体的高密度遗传图谱。图谱中27454个SNP标记整合在1554个bin中,分布在19个连锁群,总遗传距离1442.638 cM(图2)。标记之间的平均遗传距离0.928 cM。每个连锁群的平均SNP和bin标记数分别为1445和82。连锁群的遗传距离最小的为34.051 cM(LG3),最大的为107.001 cM(LG18),单个连锁群的平均遗传距离为75.928 cM。遗传图谱最大的gap出现在LG17,为15.945 cM。19个连锁群gap小于5 cM的平均占比达99.928%,其中8个连锁群占比达100%(表2)。
2.5 葡萄果实颜色性状QTL定位分析
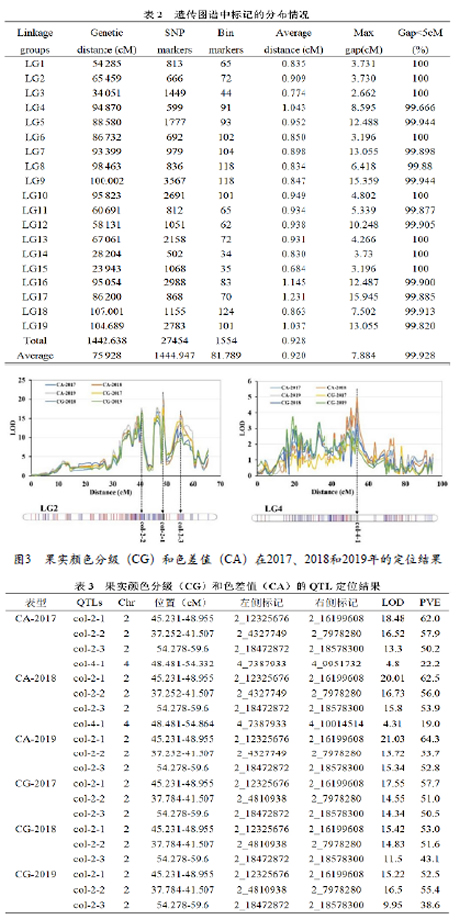
基于构建的高密度遗传图谱,结合群体果实颜色性状的表型数据(图2),进行QTL定位。果实颜色分级(CG)和色差值(CA)的定位结果较为一致,2017、2018和2019三年的表型数据均定位到LG2上的3个QTL位点,将其分别命名为col-2-1、col-2-2和col-2-3。利用CA的表型数据,在2017年和2018年还定位到位于LG4的一个QTL位点,命名为col-4-1(图3)。位点col-2-1在三年CA的表型定位中LOD值分布在18.48-21.03,解释表型变异系数(PVE)最高达64.3%(表3),表明这是一个控制葡萄果实颜色性状的主效QTL位点。此外,2017和2018年CA定位到的col-4-1位点LOD值分别为4.8和4.31,解释表型变异系数分别为19%和22.2%(表3)。
3 讨论
果实颜色作为葡萄的重要经济性状之一,其调控机理也一直是该领域研究的热点之一。葡萄果实颜色与果皮中的花色苷密切相关,位于2号染色体的MYBA1 和MYBA2 基因通过调控果皮中花色苷的积累,从而调控葡萄果实的颜色。对于葡萄果实颜色性状而言,从黄绿色系和红色系两大类着色类型考虑,果实颜色性状可以作为质量性状,MYBA1 和MYBA2 基因的同时突变导致了黄绿色果实类型的出现也很好的说明了这一问题。然而,对于红色系葡萄来说,果实颜色从粉红色到紫黑色表现出连续变异特点,符合典型的数量性状特征。
为了更好地阐释葡萄果实颜色调控机理,本领域的一些科研人员也开展了一些果实颜色相关的QTL定位研究。但是,这些QTL定位基本上都是基于由SSR标记构建的遗传图谱,受限于标记的数量,遗传图谱的密度普遍较低,直接影响了定位的精度和准确性,基本上仅定位到位于2号染色体MYBA 基因簇所在的位点。本研究中,同样定位到位于2号染色体上的该主效QTL位点(col-2-1),解释表型变异系数最高达64.3%,表明该位点的确是控制葡萄果实颜色的一个重要位点,这一结果这与前人的研究结果非常一致,这也证实了本次定位的准确性。有研究基于SSR标记和少量的SNP标记构建了一个密度较高的遗传图谱,对葡萄果实颜色性状进行QTL定位,除了定位到位于2号染色体的主效位点,还定位到了多个与葡萄果实颜色相关的QTL位点。有研究表明,提高遗传图谱的密度,能够有效提高QTL定位的精度,增加定位到微效QTL位点的可能。本研究中,基于杂交群体全基因组重测序开发的大量的SNP标记构建了高密度遗传图谱,并进行葡萄果实颜色相关性状的QTL定位,除了定位到2号染色体上的该主效QTL位点(col-2-1),在2号和4号染色体也定位到了多个与葡萄果实颜色相关的微效QTL位点。